Destacado
Científicos crearon vasos sanguíneos vivos en un chip que funcionan como los reales
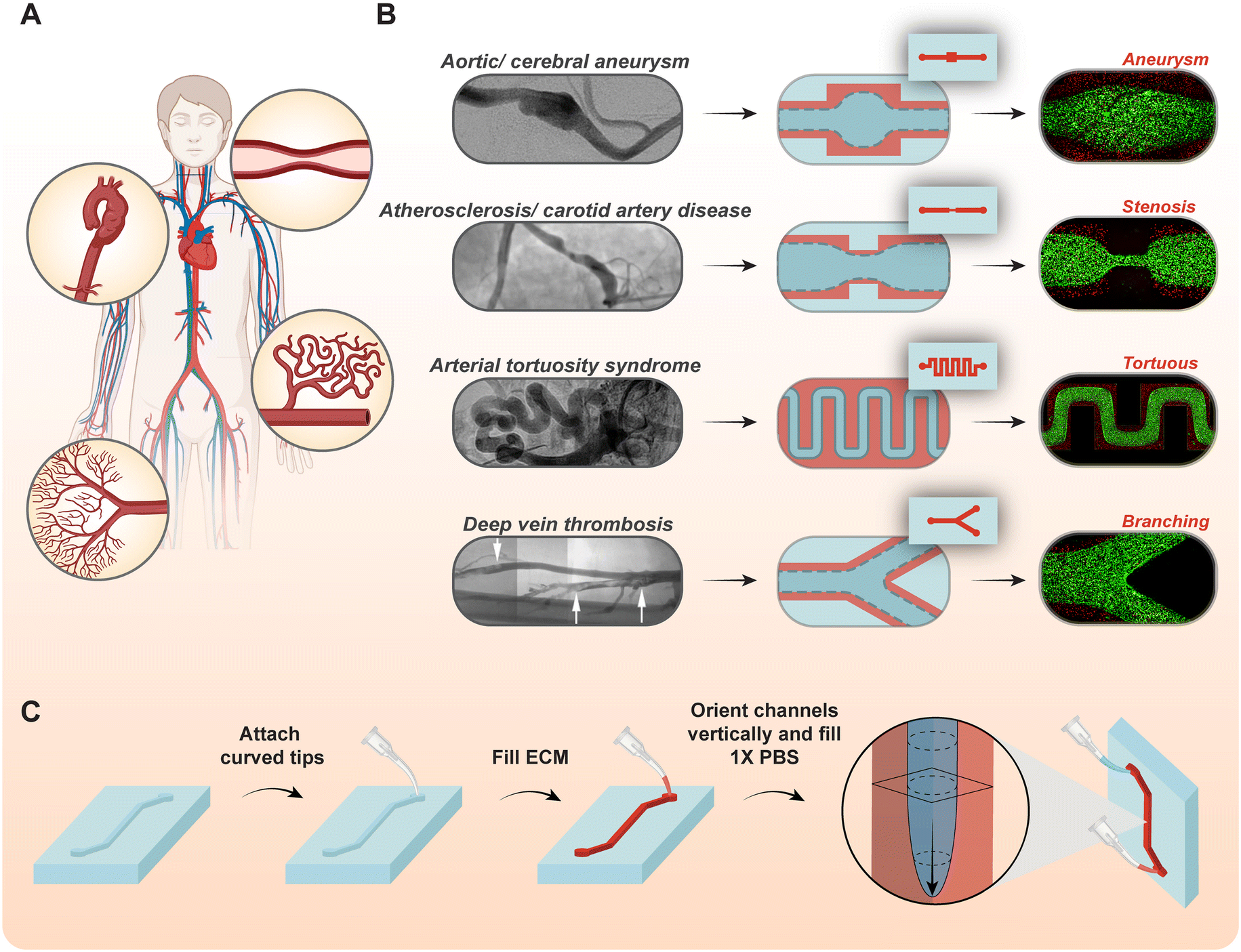
Investigadores desarrollaron vasos sanguíneos vivos en un chip microfluídico que replican comportamiento fisiológico real. El sistema reproduce fuerzas hemodinámicas y respuestas celulares, ofreciendo una plataforma avanzada para investigación cardiovascular y pruebas farmacológicas, con potencial para reducir modelos animales.
Destacado
Revisión exhaustiva de los avances metodológicos y tecnológicos en PCR durante los últimos 15 años
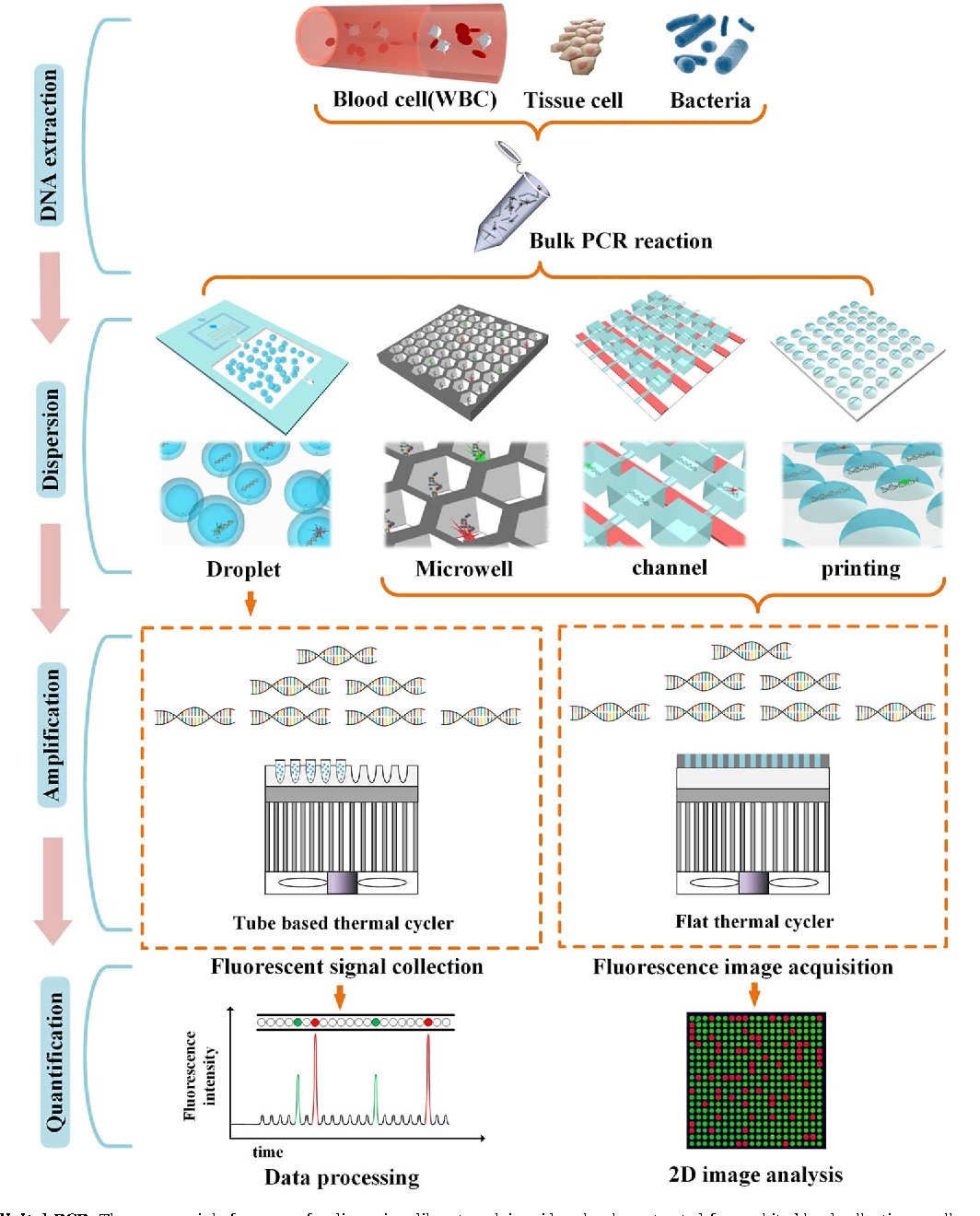
Esta revisión condensa los avances de los últimos 15 años en PCR, desde variantes como anidada, cuantitativa y multiplex, hasta versiones microfluídicas integradas en chips para diagnóstico en el punto de atención. Se destaca la transición hacia cuantificación absoluta de moléculas individuales y el uso de inteligencia artificial y visión computacional para mejorar PCR digital. El artículo proyecta que la PCR seguirá evolucionando como plataforma central en diagnóstico molecular, medición genética y medicina de precisión. https://www.sciencedirect.com/science/article/abs/pii/S0734975025002058?dgcid=rss_sd_all
Asistencia al financiamiento
Biotecnología
Maquinaria Agrícola
- Abonadoras
- Acoplados
- Arrancadoras de Papa
- Corte, tratamiento y recolección de Forraje
- Cosechadora de Algodón
- Cosechadora de Granos
- Cosechadora de Maní
- Cosechadora de Olivos
- Cosechadora de Tomates
- Cosechadoras Especiales
- Drones
- Embolsadoras
- Encaladoras
- Enfriamiento de Cereales
- Equipos de aireación
- Equipos diversos para la siembra
- Esparcidoras
- Estiercoleras
- Extractora de granos
- Fertilizadora
- Inteligencia Artificial
- Labores complementarios
- Labores extraordinarios
- Labores primarios
- Labores secundarios
- Limpiadoras y Clasificadoras de Granos
- Monitor
- Posicionamiento
- Pulverizadora
- Secadoras de Granos
- Sembradora
- Sensores
- Silos
- Sistema de guiado
- Sistema de Transporte de Granos y Mecanizaciones
- Sistemas de riego
- Tractor
- Transportador de cangilones
- Vendimiadoras


